Biologie Moléculaire et Cellulaire
Génome et Expression
1. Organisation moléculaire du Génome Eucaryote et Procaryote
Rappels sur les Acides Nucléiques
ADN = Acide DésoxyriboNucléique
Structure de l’ADN
Acide phosphorique : H3PO4
Désoxyribose : pentose en C5, le second C n’a plus d’Oxygène donc plus de groupement hydroxyle d’ou « désoxy ».
Base Purine et Pyrimidine.
Nucléoside = Ose + Base.
Nucléotide = Ose + Base + Acide P – il y a une fonction ester entre l’ose et l’acide P.
ADN = enchaînement de nucléotides monoP. L’enchaînement se fait selon une polarité entre le 5’P libre
et le 3’OH du ribose. Ainsi on établit un sens de l’ADN : 5’ -> 3’.
Conformation Générale de l’ADN en 3D = structure dite en « double hélice » => il y a un double brin. Les
deux brins correspondent avec une polarité inversée.
On note un dissymétrie au travers de l’Hélice (petit et grand sillon). On parle « d’hélice droite ».
Entre A et T = 2 liaisons hydrogènes qui stabilisent la structure ; entre G et C il y en a 3.
Le pas de l’hélice (entre les deux petits sillons) est de 33 Ansgtom (33 Å).
Il existe différentes sortes d’ADN.
-> ADN A et B : hélice droite mais A moins hydraté que le B.
-> ADN Z :hélice gauche.
Certains virus possèdent de l’ADN simple brin donc pas d’enchaînement en forme d’hélice !
L’ADN peut-être sous forme linéaire (comme toutes les cellules Eucaryotes) mais également circulaire (cf. E.coli).
Application: Cinétique de dénaturation et renaturation
L’ADN double brins absorbe à 260nm, mais il absorbe moins bien que le simple brin.
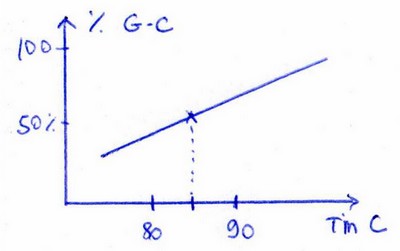
Avec Tm -> T°C de fusion de l’ADN, ~90°C. Ce Tm dépend du %G-C (Guanine – Cytosine)
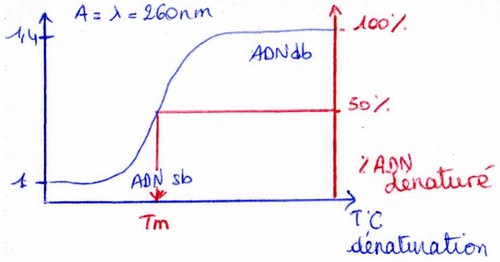
Plus il y a de G-C, plus il y a de liaison Hydrogènes, plus la dénaturation est difficile, plus il faut augmenter la Tm.
Le phénomène de renaturation est un phénomène beaucoup plus lent que la dénaturation. Il se fait à température ambiante : 20-25°C. Réaction lente car il faut que l’ADN retrouve sa conformation parfaite ave une très bonne stabilité.
Etude comparative entre génome Procaryote et Eucaryote
La structure en double hélice est la même. C’est l’organisation générale qui change.

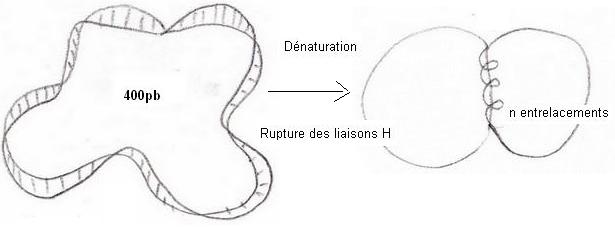
RMQ : ADN linéaire / ADN circulaire
Linéaire = 400pb = 40 enroulement donc 100pb par enroulement.
Circulaire :
Acides Nucléiques = support de l’information génétique
=> Support de caractères transmissibles ?
ADN = double brin, ARN = simple brin
Transformation bactérienne chez le pneumocoque
Il s’agit d’expérience qui date de 1928 , réalisée par Avery-Griffith pour montrer que l’ADN est le support
de l’information génétique.
Steptococcus pneumoniae forme des colonies de types S (smooth = lisses). Certains pneumocoques sont non capsulé
ce qui donne des colonies du type R (rough = rugueuses).
Les souches S peuvent produirent des anticorps d’ou S1, S2, S3, … De même pour les souches R, R1,
R2, R3,… Injectées à des souris, seules les souches S provoquent la mort des souris. La souche R n’est
pas autant virulente que S, elle ne provoque pas la mort des animaux. La capsule de S permet de mieux
résister à la réaction immunitaire, notamment à la phagocytose elle est donc plus virulente. -> dites
souches S virulentes.
Par chauffage des souches virulentes S3, ce qui les détruits. Elles sont injectés aux souris qui ne
meurent pas, il n’y a plus de virulence.
Les souches S3 sont injectées en même temps que le souches R1 (vivantes) à l’animal => la souris
meurt. Sur le lieux de l’infection on retrouve des R1 capsulées.
Hypothèse : au contact de S3, R1 peut acquérir la capacité de synthèse d’une capsule.
Expérience in vitro = même résultat.
Il y a donc transformation des coques.
Nature du facteur transformant (Avery – 1944)
Avery fait les mêmes expériences que G. Il y ajours également des enzymes connues : DNAse, la
transformation ne se produit pas.
Dans un autre mélange, il ajoute de la RNAse, il obtient les bactéries transformées.
C’est donc l’ADN qui est le support de l’information génétique.
Mélange R1 + ADN purifié de S3 => il y a transformation bactérienne.
Conclusion : l’ADN est bien le facteur qui transforme les bactéries ; ainsi c’est l’ADN qui semble porter la
capacité à synthétiser la capsule.
Mécanisme de Transformation:
L’ADN en suspension dans le mélange rentre en contact avec la paroi bactérienne. Lors de la
multiplication cellulaire, la paroi devient « compétente », cad qu’elle laisse rentrer l’ADN dans la cellule.
Ainsi elle a donc acquit les morceaux d’ADN des souches virulentes. Il faut que ces morceaux soient
insérer dans l’ADN bactérien. Il faut surtout que l’ADN porte le gène de la capsule et qu’il soit exprimé
pour que la bactérie devienne capsulée.
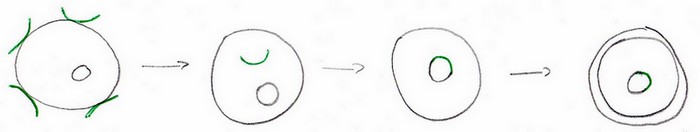
Relations entre gènes et caractères
Bactérie auxotrophe = qui a besoin d’un facteur de croissance.
Neurospora = prototrophe = souche sauvage de moisissure , Arg+ cad qui sait synthétiser de l’Arginine.
Par mutation (modification ponctuelle de l’ADN qui devient transmissible)à on peut obtenir un mutant
auxotrophe pour l’Arginine, Arg-. On a isolé 7 mutants différents, auxotrophes pour l’Arginine.
Synthèse de l’ARGININE:
Les 7 mutants auxotrophes correspondent aux 7 activités enzymatiques déficientes. Cad que soit l’enzyme n’est pas synthétisée, soit l’enzyme est non fonctionnelle.
=> on note ici qu’un gène traduit une caractère, cad une enzyme.
Myopathie de Duchenne : maladie grave qui les muscles ne se développent pas normalement. Dans les
muscles des patients il y a une protéine anormale : la « dystrophine ». Cette protéine est codée sur le
chromosome X de la paire sexuelle. On connaît exactement le locus de ce gène, locus 21 où le gène est
différent.
Chez des formes différentes de cette maladie il y a même absence de cette protéine. Ainsi un gène =
portion d’ADN qui est capable de synthétiser une protéine spécifique. La partie fonctionnelle du gène,
qui code vraiment pour la protéine s’appelle l’exon (la partie non-codante est appelée intron).
Organisation des gènes sur l’ADN

2. Réplication de l’ADN
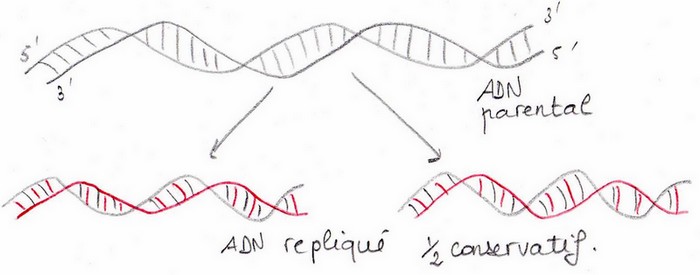
Copie d’ADN parental selon un mode ½ conservatif.
Duplication semi-conservatrice de l’ADN
Expérience de 1958, Meselson et Stahl. Expérience réalisée sur E. coli, cultivée sur un milieu 15N. On laisse se développer les bactéries pour que leur ADN ne compote que de l’azote 15N, ainsi les bases azotées sont marquées. On fait des repiquages dans des milieux contenant 14N (+ léger que le 15N). On récupère les bactéries à chaque multiplication. On extrait et on purifie leur ADN. On détecte si leur ADN comporte du 14N ou du 15N. Pour cela on fait une sédimentation de l’ADN en gradient de densité de Chlorure de Césium ( 24h – 140 000g).
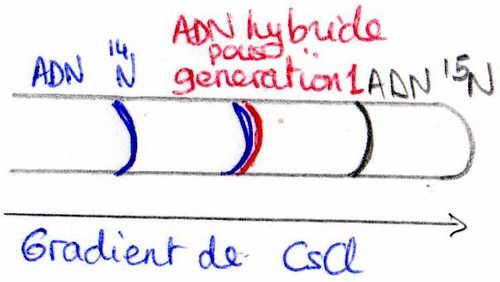
Ainsi on peut séparer les deux ADN différents suivant leur migration. Pour la première génération on ne
récupère qu’une seule bande entre 14N et 15N traduisant 100% d’ADN hybride dans deux ADN différents.
Pour la seconde génération il y a 50% d’ADN 14N et 50% d’hybride.
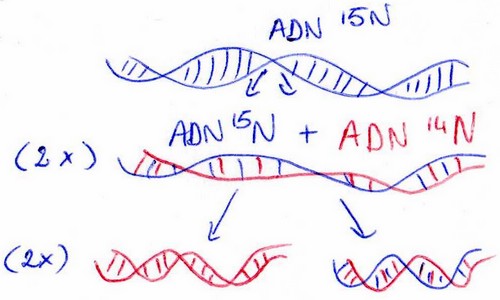
Pour la troisième génération : 25% d’hybride et 75% d’ADN 14N.
Pourquoi pas d’hybrides fragmentaires ??

On récupère la première génération que l’on dénature par un chauffage à ~85°C ce qui permet de
séparer les deux brins. Ensuite on effectue une renaturation à température plus froide. Après
renaturation, si l’hypothèse des hybrides fragmentaires est vraie alors on ne doit avoir que cette sorte
d’hybrides.. Si cette hypothèse est fausse on doit avoir des de l’ADN 14N, de l’ADN 15N ou un mélange
des deux sortes d’ADN.
Or d’après le gradient de Chlorure de Césium on obtient 3 types de bandes. Ainsi la réplication de l’ADN
n’est pas fragmentaire.
Mécanisme de la réplication ADN
Polymérisation des nucléotides
En 1955 il a été démontré que l’ADN peut-être polymérisé grâce à une enzyme = l’ADNpolymérase de
type 1. Elle a la possibilité, d’après (ADN)n, de rajouter des désoxyribonucléotides triphosphates.
Les liaisons phosphates apportent l’énergie nécessaire.
RMQ : Pour faire cette réaction il faut une « matrice ». Le plus souvent il s’agit d’ADN (double ou simple brin). D’autre part il faut également une « amorce » avec sur celle-ci l’extrémité 3’OH libre.
Enfin cette ADNpolymérase polymérise toujours 5’ -> 3’.
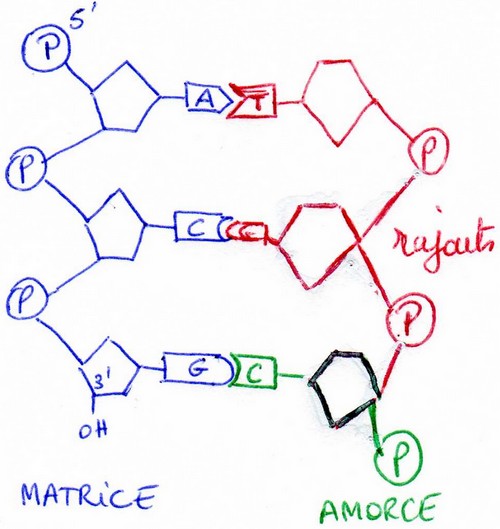
Cette enzyme utilise du Mg2+. Elle peut parfois faire des erreurs, elle a la capacité de réparer ses erreurs. Car elle a une activité exonucléasique, cad qu’elle peut couper des morceaux d’ADN. Elle coupe dans les deux sens. PM = 100Kda.
Origine de la réplication
On a noté que certains gènes sont plus répliqués que d’autres . Il y a une origine à cette réplication, c’est le site ORI.

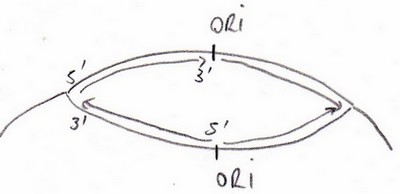
L’ADNpolymérase polymérise de 5’ -> 3’ or il y a un brin polymérisé dans l’autre sens = problème.
Chez les Eucaryotes, il existe plusieurs sites origines des réplications tous les 100 à 200 KB.
La réplication est beaucoup plus rapide chez les Eucaryotes que chez les Procaryotes (50 000 nucléotides/min pour E. ; 1 000 à 3 000 nucléotides/min pour P.).
Comme il existe une origine, il existe un arrête de la réplication 2 molécules d’ADN ½ conservatif.
Sens de la réplication de l’ADN
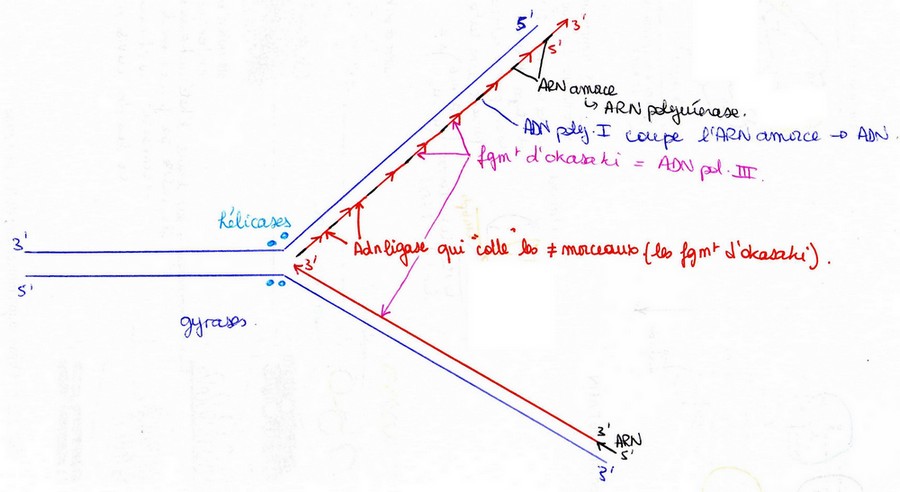
Pour la chaîne retardée, la réplication se fait par fragment, on parle de fragments d’Okasaki.
Il y a polymérisation dans le bon sens mais de tous petits fragments donc globalement avancée dans le
sens contraire. Ici c’est l’ADNpolymérase 3 qui réalise ces fragments. Elle fait également la chaîne
avancée. Ces fragments sont reliés entre eux par une autre enzyme, l’ADNligase.
Initiation de la synthèse : amorces
La réplication d’ADN commence toujours par la synthèse d’une amorce (ARN d’environ 10 nucléotides).
C’est une ARNpolymérase qui synthétise cette amorce 5’ -> 3’.
Ce qui implique que l’ARN va être hydrolysée par l’activité exonucléasique (dégradation de l’amorce).
Cette activité est portée par l’ADNpolymérase de type 1. Ainsi elle dégrade l’ARN et synthétise à
nouveau de l’ADN.
Réplisome
Association de toutes les protéines qui agissent en concert au niveau de la fourche de réplication.
Hélicases : protéine qui ouvre l’ADN en hélice. On dit qu’elle fait « fondre l’ADN ».
Gyrase : enzyme qui permet d’éviter que l’ADN se surenroule sur lui même.
RMQ : Pour E.coli on démarre la réplication à t=0min, il faut environ 20 min pour assurer la réplication
totale de l’ADN.
3. Transcription
Généralités
Passage de l’ADN en ARN. On parle notamment d’ARNmessager qui sera alors traduit en protéines.
Entre Eucaryotes et Procaryotes le phénomène est similaire. Cependant il existe des différences entre le
lieux de transcription : noyau pour E. et cytoplasme pour P.
D’autre part les ARNm des Eucaryotes subissent une « maturation » contrairement à ceux des
Procaryotes. Cad qu’ils connaissent un phénomène d’épissage : les exons codants sont conservés, les
introns non-codants ne sont pas conservés.
Conséquences de l’exportation : les ARNm des Eucaryotes sont plus stables, tps 1/2vie plus long
d’environ 1heure.
Caractères communs et différences entre la réplication et la transcription

Présentation générale des différentes étapes de la transcription
Première étape
L’ARNpolymérase se fixe sur l’ADN.
Initiation de la Transcription
Au bout d’un moment il va y avoir le début de la synthèse d’AN sur un site précis. L’ARNpolymérase a
ouvert la double hélice. La transcription commence au site +1 de l’ADN (=opérateur) du brin codant (=
matrice).L’élongation se poursuit quand ‘ARNpolymérase continue son trajet le long de l’ADN.
Il s’agit dune addition de nucléotides avec une progression extrêmement rapide ( environ 50 nucléotides
par secondes chez les Procaryotes).
La terminaison
Elle ne concerne qu’une partie de l’ADN car on ne transcrit qu’un gène. L’ARNpolymérase doit reconnaître « un site d’arrête de la transcription ». On parle de « terminateur ».
Présentation de l’ARNpolymérase (chez les Procaryotes)
Généralités
L’ARNpolymérase a besoin des 4 ribonucléotides triphosphates = ATP, UTP, GTP, CTP. Elle a
également besoin de Mg2+/Ca2+ (cations bivalents). De la même manière que l’ADNpolymérase,
l’ARNpolymérase forme des liaisons phosphodiester entre 3’OH-5’P. Elle synthétise donc dans le sens
5’ -> 3’. L’ARNpolymérase a besoin d’une matrice qui est de l’ADN double brin. L’ARNp se lie à l’ADN et
s’y déplace. Ici l’ARNp n’a pas besoin d’amorce, on dit qu’elle fonctionne « de novo ».
L’initiation de la transcription démarre généralement par une purine (A ou G).
Structure de l’ARNpolymérase
Elle est constituée de 5 chaînes peptidiques (5 sous-unités alpha, bêta, bêta prime, sigma et oméga).
On parle « d’holoenzyme ». Il s’agit d’une protéine allongée qui, en se fixant sur l’ADN se fixe sur
environ 60 nucléotides.
- sous–unité alpha : un rôle essentiellement dans la fidélité de la transcription,
- sous-unité bêta : agit lors de l’initiation est de l’élongation de la transcription. C’est elle qui créer la
liaison phosphodiester. On sait que l’antibiotique Riboficine se fixe sur cette sous-unité.
- sous-unité bêta prime : seule capable de se lier avec l’ADN. (quand il manque la sous-unité sigma il y a
une fixation non-spécifique)
- sous-unité sigma : rend la fixation de bêta prime spécifique à certains sites. Ces sites sont dits
« promoteurs », ils sont situés en amonts du site +1.
Initiation et site promoteur
Initiation
L’ARNp va se fixer sur le site promoteur de l’ADN, en amont du site +1.
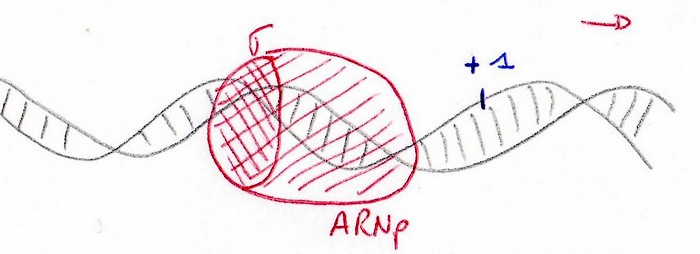
Il y a « fusion de l’ADN » = ouverture de 2 brins.
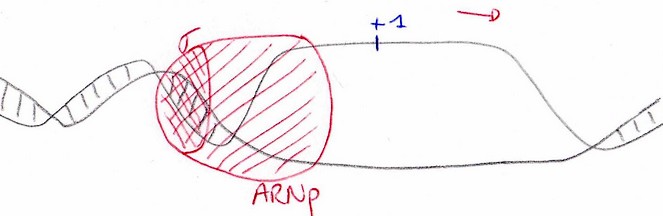
Puis l’initiation de la transcription. L’enzyme commence donc a synthétiser de l’ARN qui copie, d’après le site +1, en bases complémentaires.
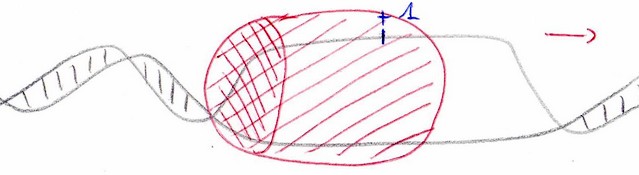
Il y a alors relarguage de la sous-unité sigma dès le début de la transcription. Il reste alors un morceau [alpha – bêta - bêta prime] dite « enzyme minimale » (ou « core enzyme »). C’est elle qui poursuite la transcription en se déplaçant sur l’ADN. L’ADN produit est le complémentaire du brin codant de l’ADN.
Séquence promoteur (chez Eucaryotes)
Elle est reconnue par l’ARNp complète (avec toutes les sous-unités). On parle d’holoenzyme. Ces
séquences sont toujours situées en amonts du site +1 ; elles sont dites « consensus », situées entre –35
et –10. Ces séquences de nucléotides sont généralement les mêmes chez les Eucaryotes à la région –
10 : TATAAT, et à la région –35 : TTGACA. Ces régions se nomment « TATAbox ». On pense que c’est
la sous-unité sigma qui reconnaît ces régions promotrices.
Il existe des « promoteurs forts » dont la séquence ne varie pas avec les régions TATAbox donc l’ARNp
reconnaît très bien ces promoteurs, l’initiation est donc plus facile.
Il existe des « promoteurs faibles » dont les séquences ne sont pas totalement comme les TATAbox,
donc al fixation de l’ARNp est moins facile.
Donc certains gènes sont plus transcrits que d’autres (fréquences d’initiation différentes si promoteurs
forts ou faibles).
Elongation et Terminaison
[alpha – bêta – bêta prime] = enzyme minimale qui se déplace sur l’ADN = élongation. L’ARN est
polymérisé dans le sens 5’ -> 3’. La vitesse d’élongation chez les Procaryotes est de 40 à 50 nucléotides
par secondes.
Il existe des signaux d’arrêt, les signaux de « terminaison ». Ce sont des structures dites palindromiques
(ou en « épingles à cheveux »).
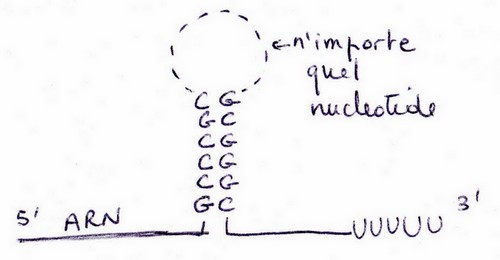
Sur le brin codant de l’ADN on trouve des régions riches en G-C puis riche en Adénine (donc U sur
l’ARN).
La molécule d’ARN va prendre une certaine structure en 3D = « épingle en cheveux ». C’est cette
structure qui traduit l’arrêt de la transcription par l’ARNp. L’ARNp se détache alrs de l’ADN. FIN de la
transcription.
RMQ : dans certains cas, la structure particulière ne suffit pas pour terminer la transcription. La protéine
Rho est alors nécessaire, on parle alors d’une « terminaison rho dépendante ». mais la protéine rho
n’est pas systématique.
Régulation de la Transcription
Il existe une régulation des gènes.
Cf. diverses fréquences d’initiation entre promoteurs forts et faibles ; cf . E. coli 4000 protéines produites
par son génome or il y en a environ que 100 qui sont produites à un moment donné. Il y a donc une
régulation de l’expression des gènes (sur la traduction et la transcription).
Cf. E. coli cultivée préférentiellement sur un milieu Glucose + Lactose (comme Kliger) : le glucose est
utilisé préférentiellement puis le lactose. On dit que les enzymes dégradant le glucose sont
« constitutives » et elles sont donc toujours produites. A contrario celles du lactoses sont « induites » car
elles ne sont produites qu’en présence du Lactose ET en absence du glucose, ces enzymes ne sont
donc pas toujours produites, elles sont donc régulées.
Opéron Lactose
Opéron = ensemble de gènes codant pour des enzymes impliquées dans l’utilisation du Lactose ici.
C’est une régulation qui concerne l’initiation de la transcription qui implique plusieurs gènes. Les
opérons sont donc inductifs. Il existe un répresseurs de l’opéron lactose qui inhibe l’initiation.
Le Lactose empêche la répression = production d ‘enzymes pour le lactose.
De nombreuses transcription nécessitent la protéine CAP.
CAP = Catabolit Activation Protein
Cette protéine peut se fixer à l’ADN pour favoriser la fixation et l’action de l’ARNp. C’est le cas pour la
transcription de l’opéron lactose : l’ADN a besoin de la protéine CAP pour transcrire l’opéron lactose. On
a remarqué qu’en présence de glucose, la concentration d’AMPc est faible. Or l’AMPc se fixe aux
protéines CAP ( [CAP-AMPc] ) et c’est cette protéine CAP-AMPc qui se fixe sur l’ADN et sert
d’activateur de l’opéron lactose. Donc en présence de Glucose la concentration d’AMPc est faible donc
la protéine CAP-AMPc ne peut pas se fixer sur l’ADN et donc il n’y a pas d’activation de la transcription
de l’opéron lactose.
Ainsi en présence ce de glucose il y a inhibition de l’utilisation du lactose. Il faut attendre que la
concentration en glucose diminue significativement afin que la concentration en AMPc augmente et que
la protéine CAP-AMPc puisse se fixer sur l’ADN. Mais il faut cependant du lactose pour que le
répresseur ne soit pas actif.
Opéron Trp
Ensemble de 5gènes important dans la biosynthèse du tryptophane. Il faut du PEP ; Phosphoénolpyruvate + érythrose 4phosphates + 5 enzymes => tryptophane.
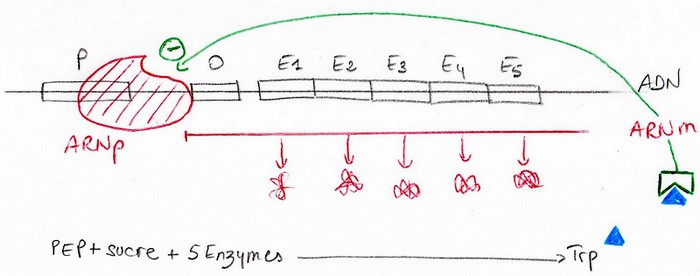
la régulation de cet opéron provient du produit final de cette biosynthèse, le Trp. Le Trp se fixe sur un récepteur qui devient alors actif. Il vient alors se fixer sur le promoteur qui empêche d’ARNp de s’y fixer. Régulation de sa propre formation.
Réponse SOS
Réponse des cellules suite à une agression. Ex : choc thermique.
Très souvent impliqué dans des mécanismes de réparation de l’ADN. Souvent après une agression il y
aune liaison de l’ADN = coupure. A la suite = blocage de la réplication. Cet arrêt de la réplication est à
l’origine d’un signal qui entraîne la transcription de la protéine RecA. Cette protéine détruit un répresseur
des enzymes du système SOS.
Transcription des gènes du système SOS qui réparent la lésion initiale.
Transcription chez les Eucaryotes
Il existe des différences essentielles avec les Procaryotes.
La Transcription se fait dans un compartiment bien définit chez les Eucaryotes = le noyau alors que la
Traduction de se fait dans le cytoplasme. Chez les Procaryotes il n’y a q’une seul ARNp, il y en a au
moins 3 de connues chez les Eucaryotes. Chacune des ces ARNp à une spécificité de synthèse (ARNr,
ARNt, ARNm).
A l’intérieur du noyau il existe différents compartiments:
- nucléole où il y a un cortex plus dense ; endroit où se situe la transcription des ARNr et ARNt.
- nucléoplasme = reste du noyau, dans chromatine, il y a transcription de l’ADN en « hnARN », « high
nuclear ARN » qui sont les pré-ARNmessagers.
Mécanisme de transcription chez les Eucaryotes:
Initiation: Nécessite des facteurs pour l’initiation de la transcription, cad des protéines qui doivent se
fixer en amont et aval du site +1. Plusieurs facteurs aident l’ARNp à commencer la transcription:
Elongation: l’ARNp migre dans le sens 5’ -> 3’.
-> faire une « coiffe » sur l’ARN qui vient juste d’être transcrit, cad addition d’un nucléotide particulier = Guanine méthylée en position 5’.
-> facteur d’élongation qui favorise d’action de l’ARNp
Terminaison: s’effectue au niveau du site « polyA » sur l’ADN, l’ARN transcrit va se détacher de l’ARNp.
Il y a alors une addition d’une séquence de plusieurs Adénine (= « polyA ») au niveau du transcrit qui
vient de se détacher.
L’ARN pré-messager va subir une maturation, c’est « l’épissage ». On observe un phénomène de
discontinuité entre l’ARNm et la séquence en AA des protéines. Dans l’ARNm il y a des parties codantes
= « exons » et des parties non-codantes = « introns ».
Mécanisme de l’épissage:
Mécanisme extrêmement complexe, il nécessite des petites particules nucléaires
« ribonucléoprotéiques » dites « snRNP ». Ces particules sont à l’origine de l’épissage, elles forment des
« lassos » qui rapprochent 2 parties de l’ARN avant éloignées. Ces snRNP coupent l’ARNm et recollent
les 2 bouts.

RMQ: après l’épissage, l’ARNm migre dans le cytoplasme.
Ces mécanismes sont les mêmes pour obtenir des ARNt et des ARNr.
Chez les Eucaryotes on note qu’un certains nombre d’ARN sont capables de faire de l’auto-épissage.
Ce qui montre que dans certains cas les ARN peuvent faire des réaction enzymatiques.
4. Traduction
Définition
Ensemble des phénomènes cellulaires qui permettent la biosynthèse de sproéine sà aprtir
d’ARNmessagers.
Comment passe-t-on d’une séquence de nucléotides à une séquence d’AA ?
Il y a besoin de ribosomes pour lire les ARNm.
Ribosome = « complexe ribonucléoprotéique » . Il utilise des ARNt couplés aux AA.
ARNt : adaptateurs
ARNt : adaptateurs de séquences nucléotidiques en séquence en AA.
ARNt : petite molécule d’ARN, d’une taille d’environ 70-90 nucléotides. Ils sont produits lors de la
transcription, tout comme l’ARN.
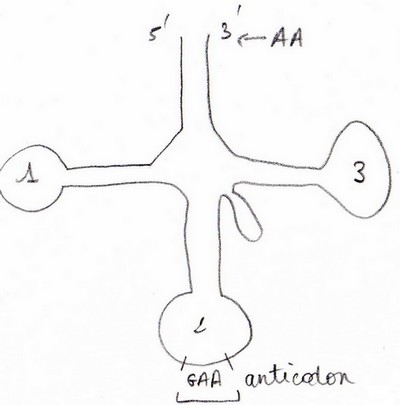
Sur l’extrémité 3’ il y a une possibilité de fixer un AA.=> un unique AA se fixe sur un unique ARNt
Il y a 31 ARNt différents dans les cellules. 1 AA pour 1 ARNt
En tout 20AA pour 31 ARNt donc 1 AA peut-être fixer par plusieurs ARNt.
L’AA se fixe par la fonction –COOH au –OH libre en 3’ = liaison éther, riche en énergie.
L’enzyme qui établit la liaison est l’anminoacylARNt synthétase.
L’AA qui se fixe sur l’ARNt dépend de l’anticodon.
La liaison entre AA et ARNt n’entraîne pas n apport complémentaire d’énergie lors de la formation de la liaison peptidique entre ARNt et AA.
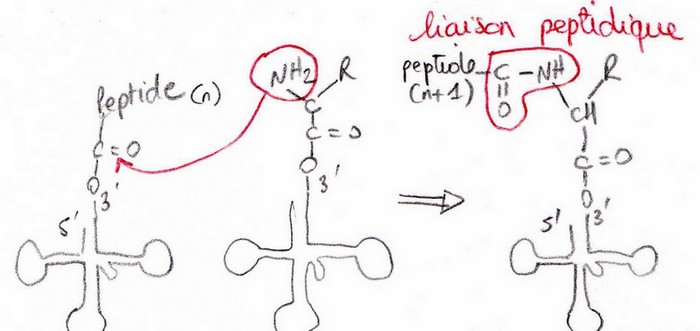
Formation d’une liaison peptidique
La formation de la liaison peptidique et la synthèse des protéines se fait progressivement. AA liés les uns après les autres.
Code Génétique
Principe de base: l'ARNm va être lu par le ribosome = appareil de traduction toujours dans le sens 5' -->
3'. La lecture se fait par groupe de trois nucléotides situés qur l'ARNm; 3 nucléotides = « un codon ». Ce
codon s'apparie avec l'anti-codon de l'ARNt. Or il y a quatre nucléotides différents, un codon = 3
nucléotides. Il y a donc 4 x 4 x 4 = 64 possibilités de codons différents. Or il y a 20 AA. Il y a 64 codons
qui codent pour 20 AA, en fait il y a 61 codons qui codent pour les AA et 3 qui codent pour l'arrêt de la
synthsèe de la protéine. On dit que el code génétique est « Dégénéré » (ou « redondant »).
3 codons stop : UAA – UAG – UGA
Avantages? La dégénescance limite les risques de changement de iosynthèse des proéines quand il y a
changement sur l'ADN ou sur l'ARNm.
Ce code génétique est universel, cad que tous els organismes vivants utilisent le même code génétique.
On note qu'il y a 61 codons pour 20 AA or il n'y a que 31 ARNt. => Certains ARNt peuvent fixer plusieurs
codons différents. Il y a donc une certaine flexibilité de reconaissance entre codns et anti-codons.
Quand on 3° position de l'ARNm on a Guanine, on peut avoir U ou C sur l'anti-codon par exemple. Il y a
une flexibilité au niveau du troisième nucléotides mais les 2 prmières positions ont une reconnaissance
structe.
Ribosomes et mécanismes de la traduction
Il n'y a pas de cheuvauchement de codons, cad que le cadre de lecture de la Traduction est 3
nucléotides par 3 nucléotides (codon par condon). Il faut que c cadre de lecture soit respecté; comment
l'appareil y parivent-il? Si il y a un saut de 1 ou 2 nucléotides le cadre de lecture est déplacé, il y a alors
modification de la séquence en AA.
Ribosome = « complexe ribonucléoprotéique » de grande taille : plusieurs millions de Daltons.
Facilement observable en microscopie, donne l'aspect granuleux du RE, mais également présent dans
le cytoplasme. Les ribosomes sont constitués d'ARN ribosomaux et de plusieurs protéines. Il est
composé de 2 sous-unités, la première, petite, se fixe à l'ARNm et ARNt. La seconde, plus grosse, est
responsable de la formation de la liaison peptidique.
Les ribosomes Eucaryoutes et Procaryotes ne sont pas les mêmes sur le plan strucutral.
Etape de la Biosynthèse
Initiation: La petite sous-unité du ribosome est capable de se fixer sur l'ARNm. Il existe deux sites, P et
A, où l'on peut fixer 2 ARNt. Le site P = peptidylARNt; site A = aminoacylARNt. Il faut démarrer la
traduction dans le bon cadre de lecture. Il existe plusieurs facteurs d'initiation qui vont aider au bond
émarrage de la traduction « IF » (initiation factors). L'ARNt initiateur code toujoruspour la Methionine. Il
possède une affinité avec l'ARNm donc il y a une liaison. Cet anti-codon va rechercher le codon de la
Met = AUG il s'y fixe alors (c'est le site P). puis la fixation d'un autre anti-codon spécifique du codon
suivant sur l'ARNm. => Liaison peptidique entre ces 2 AA.
Elongation: Au niveau du site A, qui n'est dévoilé que lorsque la grosse sous-unité du ribosome se fixe à
la petite sous-unité, il va y avoir apparition d'un codonsur lequel va se fixer un anticodon sur l'ARNt,
possédant un AA.
La peptidyltransférase est une activité contenue dans le ribosome et qui vatransférer la Met sur l'AA
suivant. Il y a alors formation d'une liaison peptidique. L'ARNt sur le site P s'en va et le ribosome se
décale de 3 nucléotides contenant les 2 AA qui se trovent alors sur le site P, le site pouvant alors
recoinnître un novuel anti-codon, etc..
5. Annexes.
Documents associés
Génome & Expressionpoly associé
Ribosomes
Traduction