Microbiologie
LA RESISTANCE AUX ANTIBIOTIQUES
Introduction
Grâce à l'emploi d'antibiotiques, on a observé une nette diminution des maladies et des
suites mortelles pour de nombreuses infections (bactériennes).
Hélas, l'introduction de chaque nouvel anti-microbien a été suivi par l'émergence de souches résistantes.
Définitions de la résistance:
-> 1 souche est résistante lorsqu'elle est capable de supporter concentration d'antibiotique
supérieure à celle atteignable in vivo;
-> 1 souche est résistante lorsqu'elle supporte concentration d'antibiotique notablement
plus importante que celle qui inhibe le développement de la majorité des souches de la
même espèce.
Jusqu'à présent, les médecins disposaient de suffisamment d'alternatives pour contourner ces souches résistantes.
A l'aube du 21ème siècle, la résistance aux antibiotiques est en train de devenir un problème majeur de santé publique, tant au sein de l'hôpital qu'en médecine de ville.
Exemple: Évolution de la résistance aux antibiotiques chez Staphylococcus aureus en milieu hospitalier:
Normalement les souches de cette espèce sont sensibles à la pénicilline, mais très rapidement dès 1950 il est apparu des souches résistantes. Cette première résistance a été
contournée en développant une nouvelle β-lactamine : la méticilline ( = Pénicilline M).
Mais il est apparu des résistants à la méticilline début des années 80, et ces derniers représentaient 20-40% des souches isolées en hôpital début '90. La seule sensibilité
connue de ces souches est alors la vancomycine (peu utilisée autrement).
A quand l'émergence d'une nouvelle résistance?
Probablement sous peu: il existe déjà des souches d'Enterococcus résistantes à vancomycine (14% en 1993) avec possibilité de transmission de cette résistance aux
Staph. ...
=> risque de développement de pathologies à Staphylococcus aureus incurables = retour à l'ère préantibiotiques !!!
Il s'agit d'une véritable course entre chercheurs et μorganismes pour découverte de nouvelles molécules actives et contournement de leur activité par résistance. Problème :
entre 10-12 ans pour découverte et 1 milliard de francs / qq années pour apparition résistance.
Découverte de nouvelles molécules de plus en plus difficile => recherche raisonnée s'appuyant sur mécanismes d'action des antibiotiques, mais également compréhension des
mécanismes de résistance + emploi réfléchi des antibiotiques.
Dans ce cours sur la résistance aux antibiotiques, nous allons développer 2 aspects principaux:
-> Origine et support génétique des résistances
-> Mécanismes biochimiques des résistances
1. D'où vient la résistance aux antibiotiques ?
Résistances naturelles / résistances acquises
Certains genres ou familles bactériens sont naturellement résistants à un antibiotique ou famille d'antibiotique donnée.
Causes:
– non pénétration de l'antibiotique
– absence de cible ou cible modifiée.
Ceci définit le spectre activité de base de l'antibiotique.
Exemples:
- les Enterobacteriaceae sont naturellement résistantes aux pénicillines traditionnelles (non diffusion de l'antibiotique à travers la membrane externe). Ona donc un développement de
l'ampicilline (=aminopénicilline), capable de traverser les membranes externes.
- Bactéries Gram+ sont naturellement résistantes à la polymyxine, car elle agit essentiellement sur la mb externe.
- Bactéries anaérobies sont naturellement résistantes aux aminosides, car ils nécessitent un transport actif à travers la mb, or l'apport d'énergie est insuffisant chez anaérobies.
A l'inverse, on va s'intéresser aux résistances acquises, apparues au sein d'espèces traditionnellement sensibles.
Exemples: - Staph cités en intro, Enterobactéries et ampicillines....
De nombreux mécanismes moléculaires sous-tendent ces résistances -> à développer + loin; d'abord développement des supports génétiques.
Déterminisme génétique de la résistance
Résistances par mutations chromosomiques
Il s'agit de modifications qui affectent des gènes déjà existant dans la bactérie.
Elles provoquent des changements de structures existantes qui rendent la cellule imperméable à un ou plusieurs antibiotiques ou qui modifient les cibles de l'antibiotique, les
rendant insensibles.
La probabilité d'apparition d'une résistance à 1 antibiotique par mutation chromosomique dépend du taux de mutations spontanées dans le génome : 10^-6 à 10^-9.
Ces mutations sont stables et héréditaires (transmissibles uniquement à la descendance).
Mais les bactéries ainsi mutées sont le plus souvent fragilisées. Elles laissent rapidement la place aux bactéries sauvages dès que les antibiotiques auxquels elles résistent
disparaissent du milieu (disparition de la pression de sélection.)
Résistances par acquisition de gènes
De nombreux gènes codant des facteurs de résistance à antibiotique se trouvent sur éléments génétiques mobiles.
Les Transposons: encadrés par deux séquences inversées répétées, capables de se copier et de se coller à différents endroits du génome bactérien.
Les Plasmides: 1 – 400kb, présents dans presque toutes les espèces bactériennes, conjugatifs (=autotransmissibles car possèdent les gènes de transfert) ou non (mais sont
alors mobilisables). Codent de nombreuses fonctions dont résistance aux antibiotiques = plasmides R ou facteurs R.
Les risques:
- transposons et plasmides conjugatifs = mobilisation vers autres souches (possibilité de transmission horizontale)
- possibilité de nombreux gènes de résistance (jusqu'à 8 gènes).
Concerne la majorité des cas isolés en clinique.
Quelques antibiotiques pour lesquels aucun transfert génétique n'a été mis en évidence: quinolones, furanes, novobiocine, polypeptides(bacitracine) rifampicine.
Transmission d'une résistance au sein d'une population
Cas des mutations chromosomiques
L'antibiotique n'est pas directement mutagène, il sélectionne les rares mutants résistants au sein d'une population sensible.
=> importance de la notion de "pression de sélection" exercée par un antibiotique donné.
Rmq: Controverse de J Cairns qui considère que l'antibiotique constitue un stress susceptible d'activer les mécanismes de mutagénèse (vrai pour antibiotique tels que
quinolones et novobiocine).
Cas de l'acquisition de gènes
Là encore: antibiotique constitue une pression de sélection et il est possible qu'il favorise les transferts génétiques.
D'où viennent les gènes?
Plusieurs hypothèses non exclusives:
- gènes de résistance seraient d'anciens "gènes de maintenance" qui produisaient des enzymes nécessaires au métabolisme et qui auraient subit une série de mutations
permettant aux enzymes d'être efficaces sur de nouveaux substrats (ie sur les antibiotiques)
- gènes de résistances proviendraient des μorganismes producteurs.
- gènes de résistance à streptomycine sont très semblables à ceux chez Streptomyces griseus (= producteur)
- il a été mis en évidence la présence d'ADN dans les antibiotiques fournis aux animaux ou aux arbres fruitiers (utilisation de préparations brutes d'antibiotiques).
2. Mécanismes biochimiques de la résistance bactérienne aux antibiotiques
Il existe 3 grands mécanismes.
Modification de la perméabilité
Mutation des porines de la membrane externe
Ce type de résistance ne se rencontre que chez les bactéries (Gram (-) et pour des
antibiotiques hydrophiles (comme les β-lactamines et aminosides, incapables de traverser
directement la membrane lipidique).
Ces mutations entraînent des modifications quantitatives ou qualitatives des porines (ex:
OmpF = outer mb prot, chez E. coli et Serratia, donnent résistances croisées).
Mutation des perméases de la membrane interne
ex : Mutation du transport actif des aminosides chez Pseudomonas aeruginosa. Mutants montrent un défaut d'énergisation oxydative de la mb = même phénomène que chez anaérobies.
Augmentation du flux de sortie
Principale résistance aux tétracyclines (antibiotique agissant sur ss-u 30s des ribosomes)
due à sortie active de l'antibiotique à travers la mb plasmique, via un anti-pore protons / tétracycline.
Ces gènes se retrouvent chez les organismes producteurs (Streptomyces).
Modification de la cible
Résistance à la méticilline
(= β lactamine) chez Staphylococcus par modification des PLP (Protéines Liant les Pénicillines).
Il a été mis en évidence, chez les Staphylococcus résistants aux β-lactamines, la présence d'un gène (mecA) codant une PLP2a, modifiée, qui présente faible affinité pour les β-
lactamines. Il confère une résistance à toutes les β-lactamines.
Ce gène mecA se trouve sur un chromosome, il est commun à tous les Staphylococcus résistants et ne présente pas d'allèles chez les souches sensibles. => idée d'un transfert
horizontal à partir autre genre.
Métabolisation de l'antibiotique
Résistance aux β-lactamines par l'acquisition de β-lactamases
Les β-lactamases sont des enzymes qui agissent sur les β-lactamines de la même façon que les transpeptidases (PLP) qui sont inhibées. La seule différence est qu'elles ne sont pas
liées de façon covalente aux antibiotiques, elles les relâchent après clivage, ce qui leur permet de rester actives. Elles sont extrêmement répandues parmi les μorganismes.
Elles illustrent bien la course à l'efficacité entre la recherche de nouveaux antibiotiques et l'apparition de résistance:
- Exemple des Staphylococcus en introduction,
- Enterobactéries:
-> elles sont naturellement résistantes à la pénicilline G mais sensibles à ampicilline.
-> puis apparition de souches résistantes à ampicilline par acquisition d'un gène de β-lactamase,
-> développement de nouvelles β-lactamines: céphalosporines de 2nde et 3ème génération,
-> apparition de nouvelles résistances par différents mécanismes:
=> Enterobacter cloacae: hyper production d'une β-lactamase chromosomique
=> Mutations ponctuelles sur 1er gène de β-lactamase étendant son spectre action (TEM-12 ...) et apparition de céphalosporinases;
=> tentative de réaction en utilisant des inhibiteurs de β-lactamases acide clavulanique), mais là encore réponse par mutation qui diminue affinité de la cible.
Résistance aux aminosides par modification de l'antibiotique
= principal mécanisme de résistance aux aminosides. Très répandu chez G+ et G-.
Il existe 3 classes d'enzymes portées par des plasmides:
- phospho-transférases : Aminoside + groupement Phosphate
- adénylyl- transférases : Aminoside + adénosine
- acétyl-transférases : Aminoside + groupement acétyl.
3. Détermination des phénotypes de résistance au laboratoire
Avec le développement des résistances acquises, il est devenu nécessaire de connaître si les souches sont résistantes ou sensibles à un antibiotique donné avant de débuter une antibiothérapie.
Evolution d'une population bactérienne en fonction de la concentration en antibiotique
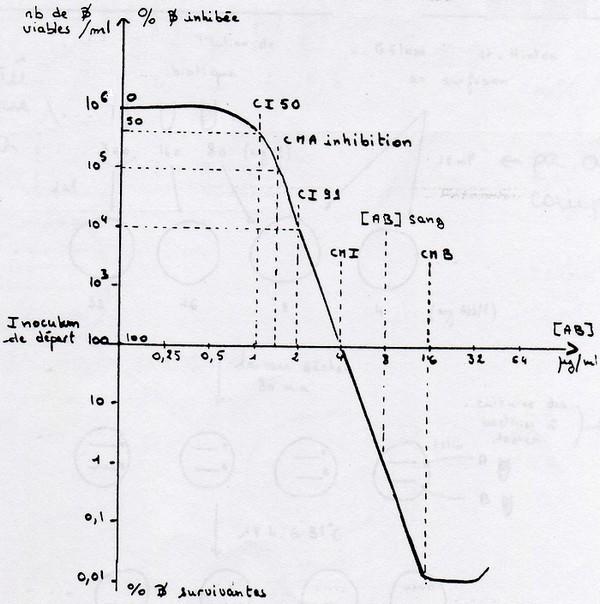
Définitions
CMI: Concentration Minimale Inhibitrice ou Concentration minimale pour laquelle en 24h
on a le même inoculum de départ, empêche la croissance des μorganismes, mais on ne les détruit pas.
CMB: Concentration Minimale Bactéricide, c'est la plus faible concentration en antibiotique
qui permet d'obtenir 0,01% de l'inoculum de départ, cela dépend de l'antibiotique et de la souche.
CI50: Concentration Inhibitrice 50%; c'est la concentration minimale en antibiotique qui
permet de limiter de 50% la croissance bactérienne (cad population bactérienne finale / 2).
CMA inhibition: Concentration minimale active en inhibition, cad la concentration
minimale en antibiotique qui permet de diminuer d'un log (cad division par 10) la population finale bactérienne.
CI99: Concentration Inhibitrice 99%; cad la concentration minimale en antibiotique qui
permet de limiter à 99% la population bactérienne finale (il ne reste qu'1% de la population finale).
Détermination de la CMI et de la CMB
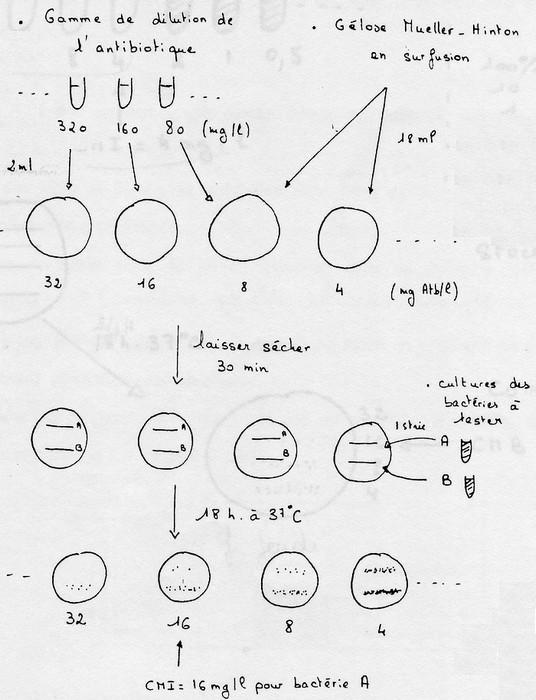
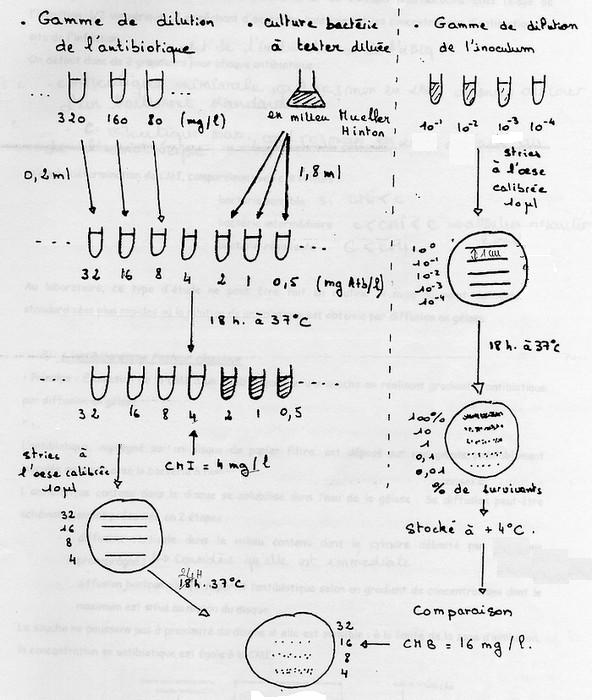
Définition clinique de la bactériostase et de la bactéricidie
In vivo, il intervient des contraintes de toxicité et de données pharmacocinétiques (siège de
l'infection, 1/2 vie sérique ...) empêchant d'accroître indéfiniment les concentrations d'antibiotique au site de l'infection.
On définit donc de 2 grandeurs pour chaque antibiotique:
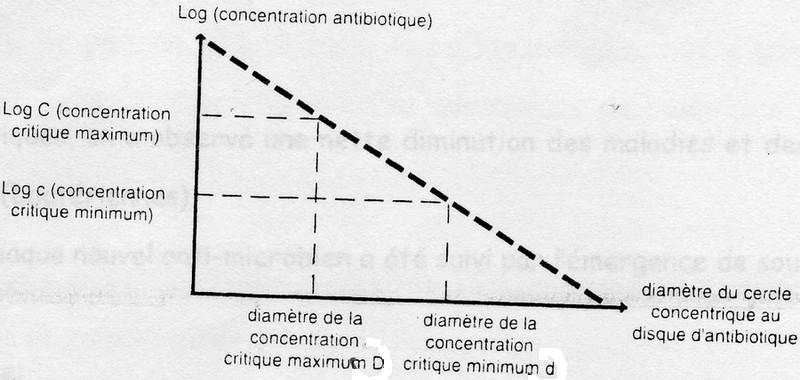
– c = « concentration critique minimale », cad concentration minimale en antibiotique obtenue au cours d'un traitement standard;
– C = « concentration critique maximale »; cad concentration maximale en antibiotique obtenue au cours de l'antibiothérapie.
=> après détermination de CMI, comparaison avec ces valeurs:
bactérie sensible si CMI < c
bactérie intermédiaire si c < CMI < C
bactérie résistante si C < CMI
Au laboratoire, ce type d'étude ne peut être fait en routine => mise au point de techniques standardisées plus rapides où la dilution de antibiotique est obtenue par
diffusion en gélose.
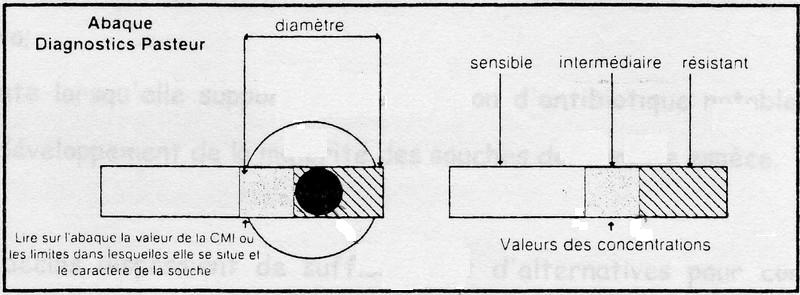
L'antibiogramme Pasteur classique
Principe: Évaluation de la CMI d'un antibiotique sur une souche en réalisant gradient
d'antibiotique par diffusion en gélose.
Technique: L'antibiotique, imprégné sur un disque de papier filtre, est déposé sur une gélose
préalablement inoculée en nappe avec la bactérie à tester.
L'antibiotique contenu dans le disque se solubilise dans l'eau de la gélose. Sa diffusion
peut-être schématiquement présentée en 2 étapes:
- diffusion verticale dans le milieu contenu dans le cylindre délimité par le disque préimprégné;
- diffusion horizontale qui répartit l'antibiotique selon un gradient de concentrations dont le
maximum est situé au niveau du disque.
La souche ne poussera pas à proximité du disque si elle est sensible : à la limite de la zone
d'inhibition, la concentration en antibiotique est égale à la CMI.
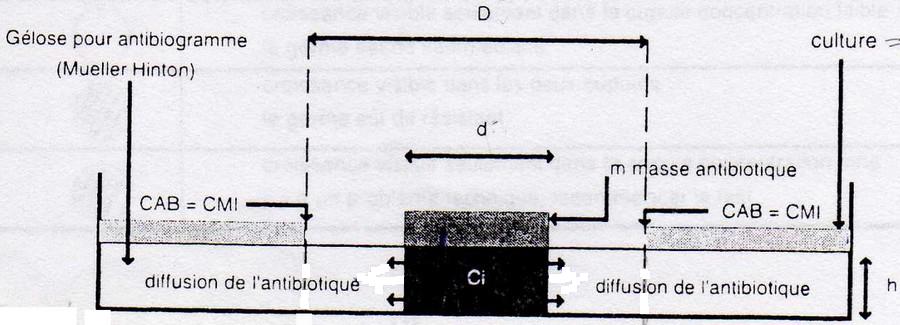
Le diamètre de la zone d'inhibition est relié au logarythme de la CMI par une relation linéaire du type:
Paramètres importants:
Nécessité de standardisation pour avoir des résultats significatifs:
- standardisation de la charge des disques en antibiotique;
- standardisation de l'épaisseur de la gélose : détermine établissement du gradient et valeur de la concentration en antibiotique à une distance donnée du disque;
- standardisation de la composition de la gélose : ex: inhibition des polymyxines par excès de cations bivalents (Ca2+, Mg2+).
- standardisation du pH : un pH trop acide augmente l'activité des p-lactamines, un milieu alcalin favorise les aminosides.
- standardisation de l'inoculum : colonies distinctes mais jointives => concentration de la culture est bien arrêtée (10^6 b/ml, d'un bouillon de 18 h).
